性别: 男
工作电话: 020-84110357
职称:副教授
电子邮件: hezuyong@mail.sysu.edu.cn
导师类型: 硕士生导师
学历: 博士
学科方向
071010-生物化学与分子生物学
0860-生物与医药
研究方向
动物基因编辑(家猪生物育种),086000 生物与医药与071010 生物化学与分子生物学专业招生。
个人经历
2016/01-至今,中山大学,生命科学学院,副教授
2013/04-2014/04,英国爱丁堡大学罗斯林研究所,国家公派访问学者
2010/07-2015/12,中山大学,生命科学学院,讲师
2008/07-2010/06,中山大学,生命科学学院,博士后,合作导师:陈瑶生教授
2003/09-2008/06,中国农业大学,生物学院,博士
讲授课程
《生物科学综合实验》- 首批国家级线下一流课程
《现代生物技术导论(核心通识)》
《大学生物实验》
学术成就
加快农业生物育种创新,是实现种业科技自立自强和种源自主可控的关键举措。本人主要围绕家猪生物育种创新开展相关研究工作,在国家自然科学基金、国家生物育种重大专项、国家转基因生物新品种培育重大专项、广东省生物育种专项、广东省重点研发项目、广东省自然科学基金和生猪养殖龙头企业横向项目等科研项目支持下,系统解析了家猪繁殖、肉质、毛色和抗病等重要经济性状的遗传调控机制,成功创制了抗病优质高产高繁殖等一批性状优异的突破性育种新素材。
1. 卵母细胞发育正常与否是影响母猪繁殖力的重要因素,通过胞质显微注射CRISPR-ctRNP的编辑技术解析猪卵母细胞体外成熟的调控机制,可为母猪繁殖调控提供坚实的理论依据。

2. 创建了双荧光筛选结合体细胞克隆法高效制作基因编辑猪的技术平台
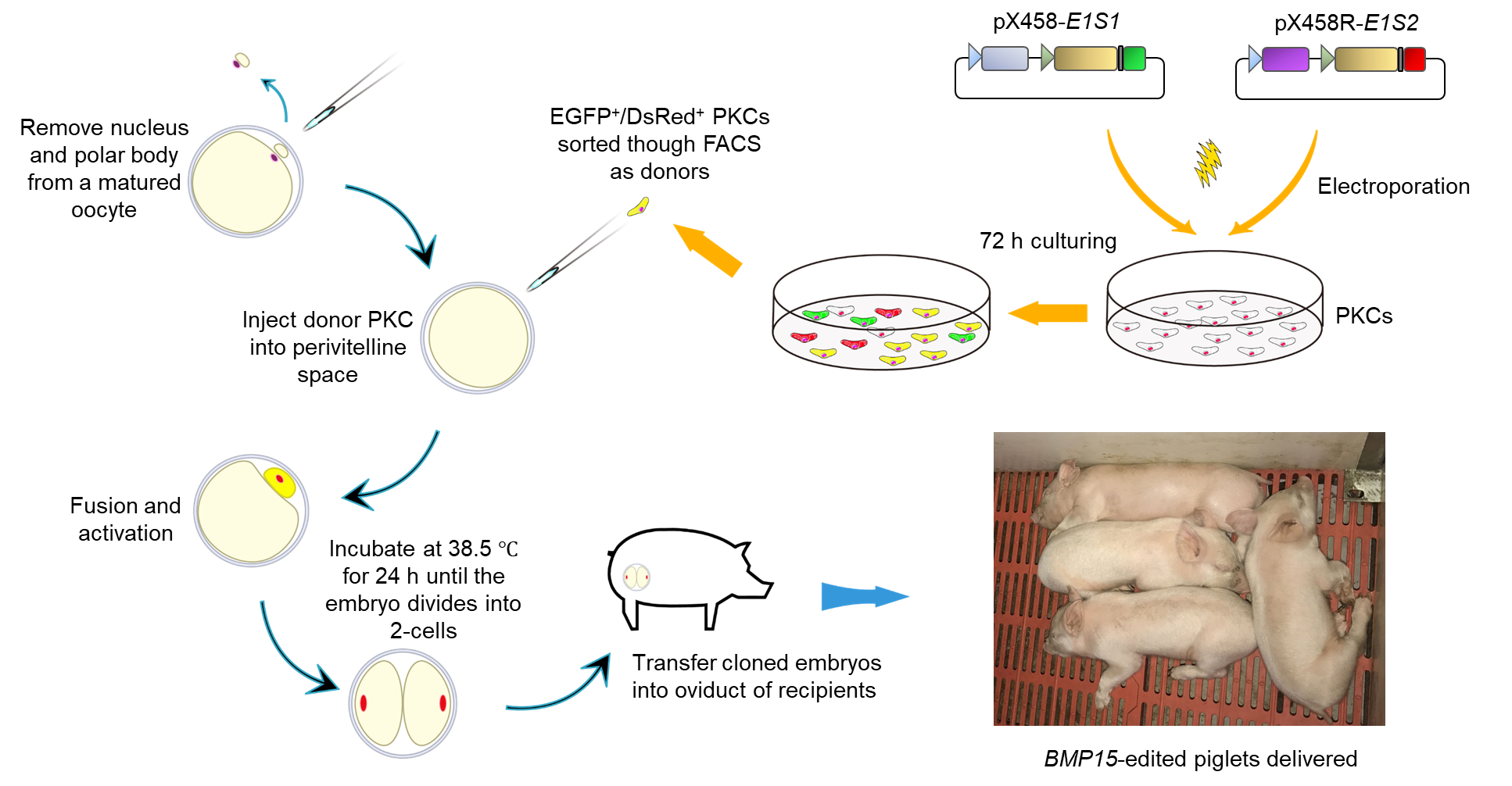
3. IGF2基因编辑两广小花猪不仅产肉量提高30%以上,肌内脂肪含量、肉色和保水性等肉质性状均获得大幅提升,打破了常规育种在追求高瘦肉率育种目标时引起肉质下降的难题,为地方猪遗传改良和生产效率提高提供了优秀范例。

4. 母猪繁殖力是决定生猪生产效率的关键因素,BMP15精准编辑杂合子母猪产仔数显著提高,加以推广应用将有效提高生猪的生产效率,创造更多的经济价值。

5. 毛色是决定家猪品种特征和商品猪销售价值的重要性状。修复KIT结构性变异的大白猪肉色性状大幅提升,并可用于杂交生产黑色优质商品猪,丰富了优质猪配套组合,具有较大的市场应用前景;MC1R基因编辑猪毛色发生显著性改变,证明其是控制家猪色素积淀的关键基因,为家猪毛色修饰提了新策略。

6. 常规育种对于抗病性新品种培育通常难以奏效,而基因编辑育种可以有效突破这种困难。CD163精准编辑猪可以完全抵抗每年给中国养猪业造成上百亿损失的蓝耳病,具有巨大的应用前景。

承担课题
- 国家自然科学基金面上项目,32573213, 2026/01-2029/12,50万,精准编辑前导肽促进猪卵母细胞BMP15蛋白分泌的调控机制研究,在研,主持
- 国家科技创新2030-农业生物育种重大项目子课题,2023ZD0404302,2023/01- 2030/12, >300万,新型抗蓝耳猪设计和研制,在研,主持
- 国家科技创新2030-农业生物育种重大项目课题,2023ZD0404706 ,2023/01- 2030/12, >500万,优质高产黑猪新品系培育,在研,参与
- 国家自然科学基金重点项目, 32030102,2021/01- 2025/12,289万,猪BMP15杂合子促进卵泡排卵及产仔数的分子机制,在研,参与
- 国家转基因重大专项子课题, 2016ZX08006003,2016/01 - 2020/12,371万,抗病高产转基因猪新品种培育,已结题,主持
- 广东省自然科学基金面上项目,2025A1515011953,2025/01-2027/12,10万,单碱基编辑IGF2降低两广小花猪背膘厚的调控机制研究,在研,主持
- 2024年省级乡村振兴战略专项资金种业振兴行动项目,2024-XPY-00-015,2025/01-2026/12,80万,种猪基因编辑技术创新及育种新材料创制,在研,子课题主持
- 广东省重点领域研发计划(现代种业)项目子课题,2018B020203003, 2019/01 – 2021/12, 243万,地方猪育种技术体系研发与新品系创建,已结题,主持
- 广东省自然科学基金面上项目,2019A1515011134,2019/10 – 2022/09,10万元,删除KIT基因剪接突变型拷贝影响大白猪造血功能的调控机制研究,已结题,主持
- 横向项目,2018-0255,2018/09 - 2023/09, 150万元, 抗蓝耳病基因编辑猪育种新材料创制,在研,主持
- 广东省自然科学基金面上项目,2016A030313310,2016/06 – 2019/06,10万元,应用CRISPR/Cas9技术删除大白猪KIT基因的冗余拷贝,已结题,主持
- 国家自然科学基金面上项目,31772565,2018/01 - 2021/12,61万元,myoD表达时间对肌纤维数量的影响及其在猪品种间表达时间差异的分子机制,已结题,参加
论文专著
1. 产肉与肉质性状遗传改良:
- Duo, T., X. Liu, D. Mo, Y. Bian, S. Cai, M. Wang, R. Li, Q. Zhu, X. Tong, Z. Liang, W. Jiang, S. Chen, Y. Chen and Z. He*(2023). "Single-base editing in IGF2 improves meat production and intramuscular fat deposition in Liang Guang Small Spotted pigs." J Anim Sci Biotechnol 14(1): 141.
- Li, R., W. Zeng, M. Ma, Z. Wei, H. Liu, X. Liu, M. Wang, X. Shi, J. Zeng, L. Yang, D. Mo, X. Liu, Y. Chen and Z. He*(2020). "Precise editing of myostatin signal peptide by CRISPR/Cas9 increases the muscle mass of Liang Guang Small Spotted pigs." Transgenic Res 29(1): 149-163.
- Liu, X., H. Liu, M. Wang, R. Li, J. Zeng, D. Mo, P. Cong, X. Liu, Y. Chen and Z. He* (2019). "Disruption of the ZBED6 binding site in intron 3 of IGF2 by CRISPR/Cas9 leads to enhanced muscle development in Liang Guang Small Spotted pigs." Transgenic Res 28(1): 141-150.
- 莫德林, 何祖勇, 彭欧阳, 童献, 王小宇, 刘小红, 曹永长,陈瑶生 (2024). "华南地方猪肉品质及抗蓝耳病机制解析和种质资源创制." 中国科学:生命科学 54(09): 1641-1650.
- 彭定威, 李瑞强, 曾武, 王敏, 石翾, 曾检华, 刘小红, 陈瑶生, 何祖勇* (2021). "编辑MSTN半胱氨酸节基元促进两广小花猪肌肉生长." 遗传 43(3): 261-270.
- 王敏, 黄翔, 石翾, 刘小凤, 曾检华, 刘小红, 陈瑶生, 何祖勇* (2017). "应用CRISPR/Cas9 编辑广东小耳花猪MSTN 基因." 广东农业科学 44(2): 141-148.
- 吴金青, 梅瑰, 刘志国, 陈瑶生, 丛佩清, 何祖勇*. (2015). 应用SSA报告载体提高ZFN和CRISPR/Cas9对猪IGF2基因的打靶效率 遗传:55-62.
- 奖励:多田琦,李瑞强,王敏,陈瑶生,刘小红,莫德林,何祖勇*(2020). IGF2基因3071位点单碱基编辑促进两广小花猪产肉量的研究. 第十七次全国畜禽遗传标记学术讨论会优秀论文奖。
- 授权专利:一种提高猪肌内脂肪含量的方法,何祖勇*; 多田琦; 刘小红; 莫德林; 陈瑶生。2025-11-14, 中国, ZL 2023 1 0982060.7。
2. 繁殖调控:
- Jiao, Y., X. Li, J. Guo, C. Bei, X. Jiang, H. Jiang, B. Mi, T. Jiang, X. Liu, Y. Chen, P. Cong and Z. He (2025). "Quercetin ameliorates oxidative stress in BMP15-deficient oocytes by regulating the ERK1/2-GSK3beta-CypD pathway." Redox Biol 87: 103886.
- Lin, S., M. Yang, W. Zhu, C. Yang, Y. Chen, P. Cong, X. Liu and Z. He (2025). "Heterozygous deletion of exon 17 of the Kit gene impairs mouse spermatogenesis by attenuating MAPK-ERK signaling." Biol Res 58(1): 28.
- Jiao, Y., A. Liao, X. Jiang, J. Guo, B. Mi, C. Bei, X. Li, T. Jiang, X. Liu, Y. Chen, P. Cong and Z. He* (2025). "Editing the growth differentiation factor 9 gene affects porcine oocytes in vitro maturation by inactivating the maturation promoting factor." Theriogenology 236: 120-136.
- Jiao, Y., Y. Zhu, J. Guo, X. Jiang, X. Liu, Y. Chen, P. Cong and Z. He* (2024). "FecB(B) mutation enhances follicle stimulating hormone sensitivity of granulosa cells by up-regulating the SMAD1/5-USF1-FSHR signaling pathway." Int J Biol Macromol 280(Pt 1): 135697.
- Jiao, Y., C. Bei, Y. Wang, A. Liao, J. Guo, X. Li, T. Jiang, X. Liu, Y. Chen, P. Cong and Z. He* (2024). "Bone morphogenetic protein 15 gene disruption affects the in vitro maturation of porcine oocytes by impairing spindle assembly and organelle function." Int J Biol Macromol 267(Pt 1): 131417.
- Jiao, Y., T. Jiang, Q. Lin, J. Guo, C. Bei, P. Cong, D. Mo, X. Liu, Y. Chen and Z. He* (2023). "Molecular characterization of the folliculardevelopment of BMP15-edited pigs." Reproduction 166(4): 247-261.
- Jiang, T., K. Wen, A. Liao, Y. Wang, Y. Jiao, J. Guo, Y. Chen, Z. He* and P. Cong (2023). "Efficient editing BMP15 in porcine oocytes through microinjection of CRISPR ctRNP." Theriogenology 198: 241-249.
- Jiao, Y., Y. Wang, T. Jiang, K. Wen, P. Cong, Y. Chen and Z. He* (2022). "Quercetin protects porcine oocytes from in vitro aging by reducing oxidative stress and maintaining the mitochondrial functions." Front Cell Dev Biol 10: 915898.
- Cai, Q., K. Wen, M. Ma, W. Chen, D. Mo, Z. He, Y. Chen and P. Cong (2021). "EZH2 is essential for spindle assembly regulation and chromosomal integrity during porcine oocyte meiotic maturation." Biol Reprod 104(3): 562-577.
- Zhang, K., P. Cong, D. Mo, Y. Chen and Z. He* (2020). "Generation and characterisation of a COV434 cell clone carrying a monoallelic FecBB mutation introduced by CRISPR/Cas9." Reproduction, Fertility and Development 32(13): 1145-1155.
- Yuan, P., L. Zheng, H. Liang, Q. Lin, S. Ou, Y. Zhu, L. Lai, Q. Zhang, Z. He* and W. Wang (2020). "Novel mutations in the PLCZ1 gene associated with human low or failed fertilization." Mol Genet Genomic Med: e1470.
- Tang, T., Q. Lin, Y. Qin, X. Liang, Y. Guo, P. Cong, X. Liu, Y. Chen and Z. He* (2020). "Effects of bone morphogenetic protein 15 (BMP15) knockdown on porcine testis morphology and spermatogenesis." Reprod Fertil Dev 32(11): 999-1011.
- Shi, X., T. Tang, Q. Lin, H. Liu, Y. Qin, X. Liang, P. Cong, D. Mo, X. Liu, Y. Chen and Z. He* (2020). "Efficient generation of bone morphogenetic protein 15-edited Yorkshire pigs using CRISPR/Cas9dagger." Biol Reprod 103(5): 1054-1068.
- Yuan, P., Z. Heco, S. Sun, Y. Li, W. Wang, X. Liang, X. Xie, Y. Jiang and D. Yang (2019). "Bi-allelic recessive loss-of-function mutations in FIGLA cause premature ovarian insufficiency with short stature." Clin Genet 95(3): 409-414.
- Qin, Y., T. Tang, W. Li, Z. Liu, X. Yang, X. Shi, G. Sun, X. Liu, M. Wang, X. Liang, P. Cong, D. Mo, X. Liu, Y. Chen and Z. He* (2019). "Bone Morphogenetic Protein 15 Knockdown Inhibits Porcine Ovarian Follicular Development and Ovulation." Front Cell Dev Biol 7: 286.
- Yuan, P., Z. Heco, L. Zheng, W. Wang, Y. Li, H. Zhao, V. W. Zhang, Q. Zhang and D. Yang (2017). "Genetic evidence of 'genuine' empty follicle syndrome: a novel effective mutation in the LHCGR gene and review of the literature." Hum Reprod 32(4): 944-953.
- 杨敏, 林思远, 杨长淇, 陈瑶生,何祖勇* (2024). "SOX9及其增强子在哺乳动物性别决定中的研究进展." 遗传: 1-17.
- 王敏, 石翾, 黄翔, 刘小凤, 覃玉凤, 刘小红, 陈瑶生, 何祖勇* (2017). "应用 RGS 双荧光替代性报告载体提高 CRISPR/Cas9 对猪 BMP15 基因的打靶效率." 遗传 39(1): 48-55.
- 覃玉凤, 李维, 陈瑶生, 何祖勇* (2017). "BMP15 基因研究进展." 农业生物技术学报 25(2): 324-334.
- 奖励:杨敏,陈瑶生,何祖勇* (2024). 删除SOX9上游增强子逆转五指山公猪性别的研究. 第十九次全国畜禽遗传标记学术讨论会优秀论文奖。
- 奖励:林思远,杨敏,陈瑶生,何祖勇*(2025). KIT外显子17杂合缺失通过削弱MAPK-ERK信号通路影响精子生成. 第二十三次全国动物遗传育种学术讨论会优秀墙报奖。
- 奖励:贺健,杨敏,陈瑶生,何祖勇*(2025). Mapping and Editing of the Gonadal Enhancer of SOX9 in Wuzhishan Pig. 第15届亚洲繁殖生物技术大会优秀墙报奖。
3. 毛色性状遗传调控:
- Zhang, C., Z. Feng, M. Yang, P. Cong, X. Liu, Y. Chen and Z. He* (2025). "Kit structural mutations associated with the porcine dominant white phenotype impair hematopoiesis in mice." FEBS Lett.
- Li, Z., J. Lan, X. Shi, T. Lu, X. Hu, X. Liu, Y. Chen and Z. He* (2024). "Whole-Genome Sequencing Reveals Rare Off-Target Mutations in MC1R-Edited Pigs Generated by Using CRISPR-Cas9 and Somatic Cell Nuclear Transfer." CRISPR J 7(1): 29-40.
- Zhang, C., M. Xu, M. Yang, A. Liao, P. Lv, X. Liu, Y. Chen, H. Liu and Z. He* (2024). "Efficient generation of cloned cats with altered coat colour by editing of the KIT gene." Theriogenology 222: 54-65.
- Liang, X., J. Lan, M. Xu, K. Qin, H. Liu, G. Sun, X. Liu, Y. Chen and Z. He* (2022). "Impact of KIT Editing on Coat Pigmentation and Fresh Meat Color in Yorkshire Pigs." CRISPR J 5(6): 825-842.
- Lan, J., M. Wang, K. Qin, X. Liu, X. Shi, G. Sun, X. Liu, Y. Chen and Z. He* (2022). "Functional characterization of cAMP signaling of variant porcine MC1R alleles in PK15 cells." Anim Genet 53(3): 317-326.
- Sun, G., X. Liang, K. Qin, Y. Qin, X. Shi, P. Cong, D. Mo, X. Liu, Y. Chen and Z. He* (2020). "Functional Analysis of KIT Gene Structural Mutations Causing the Porcine Dominant White Phenotype Using Genome Edited Mouse Models." Front Genet 11: 138.
- Qin, K., X. Liang, G. Sun, X. Shi, M. Wang, H. Liu, Y. Chen, X. Liu and Z. He* (2019). "Highly efficient correction of structural mutations of 450 kb KIT locus in kidney cells of Yorkshire pig by CRISPR/Cas9." BMC Mol Cell Biol 20(1): 4.
- 张充, 魏子璇, 王敏, 陈瑶生, 何祖勇* (2022). "利用CRISPR/Cas9在人类黑色素瘤细胞中编辑MC1R与功能分析." 遗传 44(7): 10.
- 秦珂, 黄翔, 曾检华, 王晨, 孙冠杰, 宋德清, 陈瑶生, 刘小红, 何祖勇* (2017). "陆川猪MC1R和KIT基因型鉴定." 中国畜牧兽医 44(3): 773-781.
- 秦珂, 曾检华, 薛明, 宋德清, 杨红杰, 何祖勇, 刘小红 (2017). "杜洛克×陆川猪杂交F1 代MC1R 基因型分析." 广东农业科学 44(1): 122-127.
- 授权专利:一种利用CRISPRCas9技术纠正猪KIT基因结构突变的方法,何祖勇*; 陈瑶生; 刘小红; 丛佩清; 莫德林; 刘小凤。2018-07-05, 中国, ZL201810730241.X.
4. 抗病育种:
- Guo, C., M. Wang, Z. Zhu, S. He, H. Liu, X. Liu, X. Shi, T. Tang, P. Yu, J. Zeng, L. Yang, Y. Cao, Y. Chen, X. Liu and Z. He* (2019). "Highly Efficient Generation of Pigs Harboring a Partial Deletion of the CD163 SRCR5 Domain, Which Are Fully Resistant to Porcine Reproductive and Respiratory Syndrome Virus 2 Infection." Front Immunol 10: 1846.
- 莫德林, 何祖勇, 彭欧阳, 童献, 王小宇, 刘小红, 曹永长,陈瑶生 (2024). "华南地方猪肉品质及抗蓝耳病机制解析和种质资源创制." 中国科学:生命科学 54(09): 1641-1650.
- 许美娜, 朱奕舟, 林思远, 陈瑶生, 何祖勇* (2022). "CRISPR/Cas9基因编辑技术在猪育种中的研究进展." 广东农业科学 49(8): 10.
- 奖励:中山大学-广东温氏食品集团有限公司奖励基金温氏杰出产业服务奖教金
- 奖励:第八届广东省畜牧兽医学会先进成果奖(家畜科技类):猪繁殖与呼吸综合征病毒宿主关键抗性基因解析及抗病育种新策略。
5. 基因编辑技术优化:
- Liu, X., M. Wang, Y. Qin, X. Shi, P. Cong, Y. Chen and Z. He* (2018). "Targeted integration in human cells through single crossover mediated by ZFN or CRISPR/Cas9." BMC Biotechnol 18(1): 66.
- Nie, Y., P. Cong, X. Liu, M. Wang, Y. Chen and Z. He* (2017). "Development of a bacterial-based negative selection system for rapid screening of active single guide RNAs." Biotechnol Lett 39(3): 351-358.
- He, Z., X. Shi, M. Liu, G. Sun, C. Proudfoot, C. B. Whitelaw, S. G. Lillico and Y. Chen (2016). "Comparison of surrogate reporter systems for enrichment of cells with mutations induced by genome editors." J Biotechnol 221: 49-54.
- He, Z., X. Shi, B. Du, Y. Qin, P. Cong and Y. Chen (2015). "Highly efficient enrichment of porcine cells with deletions induced by CRISPR/Cas9 using dual fluorescence selection." J Biotechnol 214: 69-74.
- He, Z., C. Proudfoot, A. J. Mileham, D. G. McLaren, C. B. Whitelaw and S. G. Lillico (2015). "Highly efficient targeted chromosome deletions using CRISPR/Cas9." Biotechnol Bioeng 112(5): 1060-1064.
- 刘小凤, 刘蔚, 聂宇, 丛佩清, 刘小红, 陈瑶生, 何祖勇* (2020). "ZFN, TALEN和CRISPR/Cas9在小鼠Rosa26基因定点整合外源基因的效率比较." 中山大学学报(自然科学版) 59(2): 137-144.
- 张楷, 刘蔚, 刘小凤, 陈瑶生, 刘小红, 何祖勇* (2019). "利用 CRISPR/Cas9 系统构建人 HPRT1 基因定点突变细胞株." 遗传 41(10): 939-949.
- 聂宇, 乔艳乐, 陈瑶生, 何祖勇* (2016). "供体同源臂长度对ZFN介导的同源重组效率的影响." 中山大学学报自然科学版 55(4): 100-107.
- 授权专利:免药物筛选快速获得小鼠Rosa26基因定点整合外源基因的方法,何祖勇*; 陈瑶生; 黄翔; 刘蔚; 聂宇; 丛佩清; 莫德林; 刘小红。2017-07-19, 中国, ZL201710590903.3.
- 授权专利:一种利用ZFN在哺乳动物细胞中实现多个大片段DNA定点整合的方法,何祖勇*; 黄翔; 刘小凤;刘小红;陈瑶生。2017-04-06, 中国, ZL201710221751.X.




